¿Qué es GLM en R?
Los modelos lineales generalizados son un subconjunto de modelos de regresión lineal y admiten distribuciones no normales de manera efectiva. Para apoyar esto, se recomienda usar la función glm (). GLM funciona bien con una variable cuando la varianza no es constante y se distribuye normalmente. Se define una función de enlace para transformar la variable de respuesta para que se ajuste al modelo apropiado. Un modelo LM se realiza tanto con la familia como con la fórmula. El modelo GLM tiene tres componentes clave llamados aleatorio (probabilidad), Sistemático (predictor lineal), componente de enlace (para la función logit). La ventaja de usar glm es que tienen flexibilidad de modelo, no necesitan varianza constante y este modelo se ajusta a la estimación de máxima verosimilitud y sus proporciones. En este tema, vamos a aprender sobre GLM en R.
Función GLM
Sintaxis: glm (fórmula, familia, datos, ponderaciones, subconjunto, Inicio = nulo, modelo = VERDADERO, método = "" …)
Aquí los tipos de familia (incluyen tipos de modelos) incluyen binomial, Poisson, gaussiano, gamma, cuasi. Cada distribución realiza un uso diferente y puede usarse tanto en clasificación como en predicción. Y cuando el modelo es gaussiano, la respuesta debería ser un número entero real.
Y cuando el modelo es binomial, la respuesta debe ser clases con valores binarios.
Y cuando el modelo es Poisson, la respuesta no debe ser negativa con un valor numérico.
Y cuando el modelo es gamma, la respuesta debe ser un valor numérico positivo.
glm.fit () - Para adaptarse a un modelo
Lrfit (): denota ajuste de regresión logística.
update (): ayuda a actualizar un modelo.
anova () - es una prueba opcional.
¿Cómo crear GLM en R?
Aquí veremos cómo crear un modelo lineal generalizado fácil con datos binarios usando la función glm (). Y al continuar con el conjunto de datos de árboles.
Ejemplos
// Importar una bibliotecalibrary(dplyr)
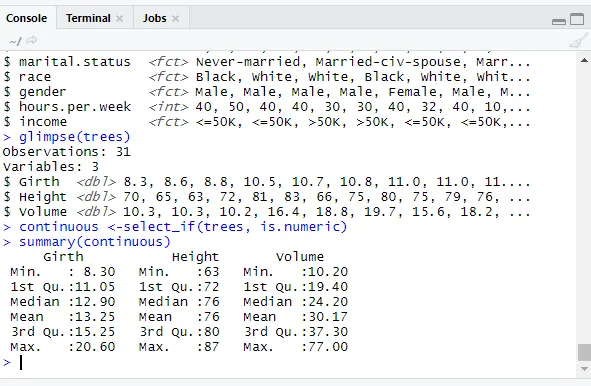
glimpse(trees)

Para ver valores categóricos se asignan factores.
levels(factor(trees$Girth))
// Verificando variables continuas
library(dplyr)
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Incluyendo el conjunto de datos de árbol en R search Pathattach (árboles)
x<-glm(Volume~Height+Girth)
x
Salida:
| Llamada: glm (fórmula = Volumen ~ Altura + circunferencia)
Coeficientes (Intercepción) Altura circunferencia -57.9877 0.3393 4.7082 Grados de libertad: 30 en total (es decir, nulo); 28 residual Desviación nula: 8106 Desviación residual: 421.9 AIC: 176.9 |
summary(x)
| Llamada:
glm (fórmula = Volumen ~ Altura + circunferencia) Residuos de desviación: Mínimo 1Q Mediano 3Q Máx. -6.4065 -2.6493 -0.2876 2.2003 8.4847 Coeficientes Estd Estd. Error t valor Pr (> | t |) (Intercepción) -57.9877 8.6382 -6.713 2.75e-07 *** Altura 0.3393 0.1302 2.607 0.0145 * Circunferencia 4.7082 0.2643 17.816 <2e-16 *** - Signif. códigos: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 '' 1 (El parámetro de dispersión para la familia gaussiana se considera 15.06862) Desviación nula: 8106.08 en 30 grados de libertad Desviación residual: 421.92 en 28 grados de libertad AIC: 176, 91 Número de iteraciones de puntuación de Fisher: 2 |
La salida de la función de resumen proporciona las llamadas, los coeficientes y los residuos. La respuesta anterior se da cuenta de que tanto el coeficiente de altura como de circunferencia no son significativos, ya que la probabilidad de que sean inferiores a 0, 5. Y hay dos variantes de desviación denominadas nulo y residual. Finalmente, la puntuación de los pescadores es un algoritmo que resuelve problemas de máxima probabilidad. Con binomio, la respuesta es un vector o matriz. cbind () se usa para unir los vectores de columna en una matriz. Y para obtener la información detallada del resumen de ajuste se utiliza.
Para hacer la prueba Like hood se ejecuta el siguiente código.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
Ajuste del modelo
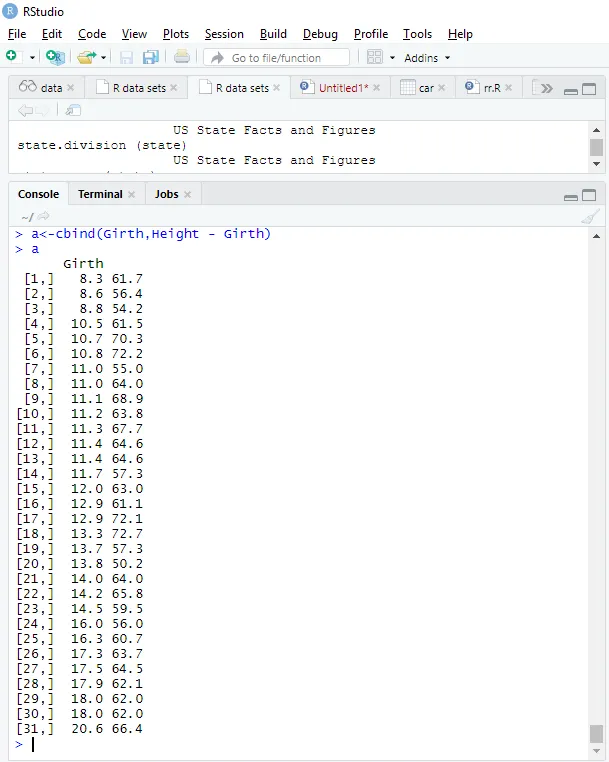
a<-cbind(Height, Girth - Height)
> a
resumen (árboles)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
Para obtener la desviación estándar adecuada
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
A continuación, nos referimos a la variable de respuesta de conteo para modelar un buen ajuste de respuesta. Para calcular esto, utilizaremos el conjunto de datos USAccDeath.
Ingresemos los siguientes fragmentos en la consola R y veamos cómo se realiza el recuento de años y el cuadrado del año en ellos.
data("USAccDeaths")
force(USAccDeaths)
// Analizar el año de 1973-1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Llamada:
glm (fórmula = cuenta ~ año + añoSqr, familia = "poisson", datos = disco) Residuos de desviación: Mínimo 1Q Mediano 3Q Máx. -22.4344 -6.4401 -0.0981 6.0508 21.4578 Coeficientes Estd Estd. Error z valor Pr (> | z |) (Intercepción) 9.187e + 00 3.557e-03 2582.49 <2e-16 *** año -7.207e-03 2.354e-04 -30.62 <2e-16 *** añoSqr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. códigos: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 '' 1 (El parámetro de dispersión para la familia Poisson se considera 1) Desviación nula: 7357.4 en 71 grados de libertad Desviación residual: 6358.0 en 69 grados de libertad AIC: 7149.8 Número de iteraciones de puntuación de Fisher: 4 |
Para verificar el mejor ajuste del modelo, se puede usar el siguiente comando para encontrar
Los residuos para la prueba. Del resultado a continuación, el valor es 0.
1 - pchisq(deviance(a1), df.residual(a1))
Usando la familia QuasiPoisson para la mayor varianza en los datos dados
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Llamada:
glm (fórmula = cuenta ~ año + añoSqr, familia = "cuasipoisson", datos = disco) Residuos de desviación: Mínimo 1Q Mediano 3Q Máx. -22.4344 -6.4401 -0.0981 6.0508 21.4578 Coeficientes Estd Estd. Error t valor Pr (> | t |) (Intercepción) 9.187e + 00 3.417e-02 268.822 <2e-16 *** año -7.207e-03 2.261e-03 -3.188 0.00216 ** añoSqr 8.841e-05 3.095e-05 2.857 0.00565 ** - (El parámetro de dispersión para la familia de cuasipoisson se considera 92.28857) Desviación nula: 7357.4 en 71 grados de libertad Desviación residual: 6358.0 en 69 grados de libertad AIC: NA Número de iteraciones de puntuación de Fisher: 4 |
La comparación de Poisson con el valor binomial de AIC difiere significativamente. Se pueden analizar por precisión y relación de recuperación. El siguiente paso es verificar que la varianza residual sea proporcional a la media. Luego podemos trazar usando la biblioteca ROCR para mejorar el modelo.
Conclusión
Por lo tanto, nos hemos centrado en un modelo especial llamado modelo lineal generalizado que ayuda a enfocar y estimar los parámetros del modelo. Es principalmente el potencial para una variable de respuesta continua. Y hemos visto cómo glm se adapta a los paquetes integrados de R. Son los enfoques más populares para medir datos de conteo y una herramienta robusta para las técnicas de clasificación utilizadas por un científico de datos. El lenguaje R, por supuesto, ayuda a realizar funciones matemáticas complicadas
Artículos recomendados
Esta es una guía de GLM en R. Aquí discutimos la función GLM y cómo crear GLM en R con ejemplos de conjuntos de datos de árbol y resultados. También puede consultar el siguiente artículo para obtener más información:
- Lenguaje de programación R
- Arquitectura de Big Data
- Regresión logística en R
- Empleos de Big Data Analytics
- Regresión de Poisson en R | Implementando la regresión de Poisson